 |
 |
- Search
| Korean J Helicobacter Up Gastrointest Res > Volume 20(3); 2020 > Article |
|
Abstract
Owing to advancements in next-generation sequencing and non-culture-based microbial research techniques, we have recognized that many bacterial taxa other than Helicobacter pylori (H. pylori) are present in the human stomach. Gastric microbial composition depends on gastric diseases, including gastritis, atrophic gastritis, intestinal metaplasia, and gastric cancer. Although H. pylori is a major factor associated with gastric cancer development, other bacterial taxa may affect gastric carcinogenesis. Because the risk of gastric cancer development can be reduced through H. pylori eradication, many investigators have studied the changes in the microbial composition in the stomach after H. pylori eradication. The gastric microbiome in patients with H. pylori infection typically shows abundance of H. pylori and a low microbial diversity index. If we treat H. pylori-infected patients with antibiotics, microbial diversity increases, and the relative abundance also increases in many bacterial taxa. Several studies suggested that the microbial composition in patients with H. pylori infection could be restored by H. pylori eradication therapy; however, there have been inconsistent findings of the abundant bacterial taxa after H. pylori eradication in patients with atrophic gastritis and intestinal metaplasia. More studies are required to reach a definitive conclusion on restoration of the microbial composition after H. pylori eradication according to the severity of gastric inflammation.
1980ļģäļīĆ ņ┤ł, MarshallĻ│╝ Warren [1]ņØ┤ ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼Ļ░Ć ņ£äņŚ╝ņØś ņøÉņØĖņ×äņØä ļ░ØĒ×ī ņØ┤Ēøä, Ēśäņ×¼Ļ╣īņ¦Ć ņ£äĻČżņ¢æ, ņŗŁņØ┤ņ¦ĆņןĻČżņ¢æ, ņ£ä ņĀÉļ¦ēĻ┤ĆļĀ©ļ”╝ĒöäņĪ░ņ¦ü(mucosa-associated lymphoid tissue) ļ”╝Ēöäņóģ ļō▒ ļŗżņ¢æĒĢ£ ņ£ä, ņŗŁņØ┤ņ¦Ćņן ņ¦łĒÖśĻ│╝ ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ņØś ņŚ░Ļ┤Ćņä▒ņØ┤ ņĢīļĀżņĪīļŗż[2]. ĒŖ╣Ē׳ ņŚ¼ļ¤¼ ņŚŁĒĢÖ ņŚ░ĻĄ¼ņŚÉņä£, ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ņÖĆ ņ£äņĢöĻ│╝ņØś ļÜ£ļĀĘĒĢ£ ņŚ░Ļ┤Ćņä▒ņØ┤ ļ░ØĒśĆņ¦Ćļ®┤ņä£ 1994ļģäņŚÉ ņäĖĻ│äļ│┤Ļ▒┤ĻĖ░ĻĄ¼ ņé░ĒĢś International Agency for Research on CancerņŚÉņä£ ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ļź╝ ņĀ£1ĻĄ░ ļ░£ņĢöļ¼╝ņ¦łļĪ£ ĻĘ£ņĀĢĒĢśņśĆļŗż[3]. ĻĘĖļ¤¼ļéś ņ£äņĢö ļ░£ņāØņŚÉļŖö ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ņÖĖņŚÉ ņŚ░ļĀ╣, ņä▒ļ│ä, ĒØĪņŚ░, ņ£äņĢö Ļ░ĆņĪ▒ļĀź, ĒøłņĀ£ Ēś╣ņØĆ ņĀłņØĖ ņØīņŗØ, ļé«ņØĆ ņé¼ĒÜīĻ▓ĮņĀ£ņĀü ņłśņżĆ ļō▒ ņŚ¼ļ¤¼ ņØĖņ×ÉĻ░Ć Ļ┤ĆņŚ¼ĒĢśļ®░[4], ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ņ×Éņ▓┤ļŖö ņ£äņĢöņØä ņØ╝ņ£╝ĒéżļŖö ņ¦üņĀæņĀüņØĖ ņøÉņØĖņØ┤ ņĢäļŗÉņ¦ĆļÅä ļ¬©ļźĖļŗżļŖö ņØśĻ▓¼ļÅä ņĀ£ņŗ£ļÉśĻ│Ā ņ׳ļŗż[5,6].
ņØ┤ņŚÉ ļö░ļØ╝ ļ¦ÄņØĆ ņŚ░ĻĄ¼ņ×ÉļōżņØ┤ ņ£äņĢöņØś ļśÉ ļŗżļźĖ ņøÉņØĖņØä ĻĘ£ļ¬ģĒĢśļĀżĻ│Ā ņŗ£ļÅäĒĢśĻ│Ā ņ׳ņ£╝ļ®░, ņØ┤ ņżæ Ļ░Ćņן ļīĆĒæ£ņĀüņØĖ Ļ▓āņØ┤ ņ£ä ļé┤ ļ»ĖņāØļ¼╝ĻĄ░ņ¦æ(microbiota)ņØ┤ļŗż[7]. ļ»ĖņāØļ¼╝ņŚÉ ļīĆĒĢ£ ņŚ░ĻĄ¼ļŖö ņĀäĒåĄņĀüņ£╝ļĪ£ 100ļģä ļäśĻ▓ī ļ░░ņ¢æļ▓ĢņØä ĒåĄĒĢ┤ ņØ┤ļŻ©ņ¢┤ņĪīņ£╝ļéś, 2000ļģäļīĆ ņ┤łļ░ś ņ░©ņäĖļīĆ ņŚ╝ĻĖ░ņä£ņŚ┤ļČäņäØļ▓Ģ(next-generation sequencing)ņØ┤ ļÅäņ×ģļÉśļ®┤ņä£ Ēśäņ×¼ļŖö ļ»ĖņāØļ¼╝ņØś DNA ņä£ņŚ┤ ļČäņäØņØä ĻĖ░ļ░śņ£╝ļĪ£ ĒĢ£ ļ╣äļ░░ņ¢æļ▓ĢņØ┤ ļ»ĖņāØļ¼╝ ņŚ░ĻĄ¼ ļ░®ļ▓Ģņ£╝ļĪ£ ļäÉļ”¼ ĒÖ£ņÜ®ļÉśĻ│Ā ņ׳ļŗż[7,8]. Ēśäņ×¼ļŖö, ņ£ä ļé┤ņŚÉļÅä ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ņÖĖņŚÉ ņŚ¼ļ¤¼ ļ»ĖņāØļ¼╝ņØ┤ ņāüņ×¼ĒĢśĻ│Ā ņ׳ņØīņØä ņĢīĻ│Ā ņ׳ņ£╝ļ®░, ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ņÖĖņŚÉ ņ£äņĢöĻ│╝ ņŚ░Ļ┤ĆļÉ£ ļ░ĢĒģīļ”¼ņĢäļź╝ ĻĘ£ļ¬ģĒĢśĻ│Āņ×É ĒĢśļŖö ļģĖļĀźņØ┤ ņ׳ņŚłļŗż[8,9]. ĒŖ╣Ē׳, ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ņĀ£ĻĘĀ ņ╣śļŻī Ēøä ļ│ĆĒÖöĒĢśļŖö ņ£ä ļé┤ ļ»ĖņāØļ¼╝ĻĄ░ņ¦æņØś ĒŖ╣ņä▒ņØä ĒīīņĢģĒĢśļĀżļŖö ņŚ░ĻĄ¼Ļ░Ć ļŗżņłś ņ¦äĒ¢ēļÉśņŚłļŗż[6,10-16]. ņØ┤ņŚÉ ļ│ĖĻ│ĀņŚÉņä£ļŖö ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ņĀ£ĻĘĀ ņ╣śļŻī ņĀä/Ēøä ņ£ä ļé┤ ļ»ĖņāØļ¼╝ĻĄ░ņ¦æņØä ĒŖ╣ņä▒ņØä ļ╣äĻĄÉĒĢ┤ļ│┤Ļ│Ā ņ£äņČĢņä▒ņ£äņŚ╝ ļ░Å ņןņāüĒö╝ĒÖöņāØ, Ēś╣ņØĆ ņ£äņĢöņØś ļ░£ļ│æĻ│╝ ņŚ░Ļ┤ĆļÉśļŖö ļ░ĢĒģīļ”¼ņĢäņŚÉ ļīĆĒĢ┤ ņé┤ĒÄ┤ļ│┤Ļ│Āņ×É ĒĢ£ļŗż.
ļ»ĖņāØļ¼╝ĻĄ░ņ¦æņØ┤ ņ¦łļ│æņŚÉ ļ»Ėņ╣śļŖö ņśüĒ¢źņØä ņØ┤ĒĢ┤ĒĢĀ ļĢī Ļ░Ćņן ņżæņÜöĒĢ£ Ļ▓ā ņżæ ĒĢśļéśļŖö ĒŖ╣ņĀĢ ĒÖśĻ▓ĮņŚÉ ņĪ┤ņ×¼ĒĢśļŖö ļ»ĖņāØļ¼╝ņØś ļŗżņ¢æņä▒ņØ┤ļŗż[17]. ņןļé┤ ļ»ĖņāØļ¼╝ĻĄ░ņ¦æņØś ļŗżņ¢æņä▒ņØĆ Ļ▒┤Ļ░ĢņØĖļ│┤ļŗż ņŚ╝ņ”Øņä▒ ņןņ¦łĒÖś ĒÖśņ×Éļéś Clostridioides difficle ņŚ░Ļ┤Ć ņןņŚ╝ ĒÖśņ×ÉņŚÉņä£ ļŹö ļé«ņ£╝ļ®░, ņØ╝ļ░śņĀüņ£╝ļĪ£ ņ¦łļ│æņØ┤ ļ░£ņāØĒĢĀ Ļ▓ĮņÜ░ ļ»ĖņāØļ¼╝ĻĄ░ņ¦æņØś ļŗżņ¢æņä▒ņØ┤ ļé«ņĢäņ¦ĆļŖö Ļ▓āņ£╝ļĪ£ ņĢīļĀżņĀĖ ņ׳ļŗż[17,18]. ĻĘĖļ¤¼ļéś ņäĖĻĘĀņä▒ ņ¦łņŚ╝ ĒÖśņ×ÉņØś ņ¦ł ļé┤ ļ»ĖņāØļ¼╝ĻĄ░ņ¦æņØĆ Ļ▒┤Ļ░ĢņØĖņØś ņ¦ł ļé┤ ļ»ĖņāØļ¼╝ĻĄ░ņ¦æņŚÉ ļ╣äĒĢ┤ ņśżĒ׳ļĀż ļŗżņ¢æņä▒ņØ┤ ļåÆĻĖ░ ļĢīļ¼ĖņŚÉ ļŗżņ¢æņä▒ņØś ņ”ØĻ░ĆĻ░Ć ļ░śļō£ņŗ£ Ļ▒┤Ļ░ĢĒĢ£ ņāüĒā£ļź╝ ņØśļ»ĖĒĢśļŖö Ļ▓āņØĆ ņĢäļŗłļŗż[19].
ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ņŚÉ Ļ░ÉņŚ╝ļÉśņ¦Ć ņĢŖņØĆ Ļ▒┤Ļ░ĢņØĖņØś ņ£ä ļé┤ ļ»ĖņāØļ¼╝ĻĄ░ņ¦æņŚÉļŖö Firmicutes, Proteobacteria, Cyanobacteria, Bacteroidetes, Actinobacteria, Acidobacteria ļō▒ ļŗżņ¢æĒĢ£ phylumņØ┤ Ļ┤Ćņ░░ļÉśļ®░, ļŗżņ¢æņä▒ ņŚŁņŗ£ ļåÆņØĆ ņłśņżĆņØ┤ļŗż[11,13]. ļ░śļ®┤, ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ņŚÉ Ļ░ÉņŚ╝ļÉ£ ĒÖśņ×ÉņØś Ļ▓ĮņÜ░ ņ£ä ļé┤ ļ»ĖņāØļ¼╝ĻĄ░ņ¦æ ņżæ ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ņØś ņāüļīĆ ĒÆŹļČĆļÅä(relative abundance)ļŖö 90% ņØ┤ņāüņØ┤ļ®░[13], ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ņØś ņāØĒā£ņĀü ņ¦Ćņ£äĻ░Ć ņś¼ļØ╝Ļ░ÉņŚÉ ļö░ļØ╝ ņ×Āņ×¼ņĀüņ£╝ļĪ£ ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ņÖĆ Ļ▓Įņ¤üĒĢĀ ņłś ņ׳ļŖö ļŗżļźĖ ļ»ĖņāØļ¼╝ņØś ņä£ņŗØ Ļ░ĆļŖźņä▒ņØ┤ ņżäņ¢┤ļōżĻ│Ā, Ļ▓░Ļ│╝ņĀüņ£╝ļĪ£ ņ£ä ļé┤ ļ»ĖņāØļ¼╝ĻĄ░ņ¦æņØś ļŗżņ¢æņä▒ņØĆ ļ¦żņÜ░ ļé«ņĢäņ¦äļŗż[11,15]. ĻĘĖļ¤¼ļéś ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ Ļ░ÉņŚ╝ņØ┤ ņ¦ĆņåŹļÉ©ņŚÉ ļö░ļØ╝ ņ£äņČĢņä▒ ņ£äņŚ╝ ļ░Å ņןņāüĒö╝ĒÖöņāØņØ┤ ņ¦äĒ¢ēļÉśļ®┤ ņ£äņé░ ļČäļ╣äĻ░Ć ņżäņ¢┤ļōżļ®░ ņ£ä ļé┤ ļ»ĖņāØļ¼╝ĻĄ░ņ¦æņØś ņĪ░ņä▒ņØ┤ ļ│ĆĒÖöĒĢ£ļŗż. ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ņØś ņāüļīĆ ĒÆŹļČĆļÅäļŖö ņżäņ¢┤ļōżĻ│Ā, non-Helicobacter Proteobacteriaļź╝ ļ╣äļĪ»ĒĢśņŚ¼ ņŚ¼ļ¤¼ ļ░ĢĒģīļ”¼ņĢä ļČäļźśĻĄ░(taxa)ņØ┤ ļŖśņ¢┤ļéśļ®░, ļ»ĖņāØļ¼╝ĻĄ░ņ¦æņØś ļŗżņ¢æņä▒ņØ┤ ļåÆņĢäņ¦äļŗż[11,13,15]. ņ”ē, ņ£ä ļé┤ ļ»ĖņāØļ¼╝ĻĄ░ņ¦æņØĆ ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ļ»ĖĻ░ÉņŚ╝ ņāüĒā£ņŚÉņä£, ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ Ļ░ÉņŚ╝, ņ£äņČĢņä▒ ņ£äņŚ╝, ņןņāüĒö╝ĒÖöņāØņØ┤ ņ¦äĒ¢ēļÉ©ņŚÉ ļö░ļØ╝ ļŗżņ¢æņä▒ņØ┤ ņżäņ¢┤ļōżņŚłļŗżĻ░Ć ļŗżņŗ£ ļŖśņ¢┤ļéśļŖö ļÅÖņĀü ļ│ĆĒÖöļź╝ Ļ▓¬Ļ▓ī ļÉ£ļŗż.
ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ņĀ£ĻĘĀ ņ╣śļŻī ņŗ£ ņ£ä ļé┤ ļ»ĖņāØļ¼╝ĻĄ░ņ¦æņØś ļŗżņ¢æņä▒ņØ┤ ļ│ĄņøÉļÉ£ļŗżļŖö Ļ▓āņØĆ ņŚ¼ļ¤¼ ņŚ░ĻĄ¼ņŚÉņä£ ļ░ØĒśĆņĀĖ ņ׳ļŗż(Table 1). ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ņĀ£ĻĘĀ ņ╣śļŻī Ēøä ņ£ä ļé┤ ļ»ĖņāØļ¼╝ņØś ļ│ĆĒÖöļź╝ ļ╣äļ░░ņ¢æļ▓Ģņ£╝ļĪ£ ņ▓śņØī ĒÖĢņØĖĒĢ£ Ļ▓āņØĆ 2016ļģä ļ░£Ēæ£ļÉ£ Li ļō▒[10]ņØś ņŚ░ĻĄ¼ļĪ£, ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ Ļ░ÉņŚ╝ņØ┤ ļÅÖļ░śļÉ£ ņŗŁņØ┤ņ¦ĆņןĻČżņ¢æ ĒÖśņ×É 21ļ¬ģņŚÉ ļīĆĒĢ┤ ņĀ£ĻĘĀ ņ╣śļŻī ņĀä/Ēøä, 16S rRNA ņ£ĀņĀäņ×ÉņŚÉ ļīĆĒĢ┤ ņĀĢļ¤ē ņŗżņŗ£Ļ░ä ņżæĒĢ®ĒÜ©ņåī ņŚ░ņćäļ░śņØæļ▓Ģ(quantitative real-time polymerase chain reaction)ņØä ņŗ£Ē¢ēĒĢśņŚ¼ ņ£ä ļé┤ ņŻ╝ņÜö ļ»ĖņāØļ¼╝ņØś ņ¢æņØä ņĖĪņĀĢĒĢśņśĆļŗż. ņĀ£ĻĘĀ ņ╣śļŻī Ēøä, ņ£ä ņĀäņĀĢļČĆņŚÉņä£ Lactobacillus, Clostridium leptum, EnterobacteriaņØś ĒÆŹļČĆļÅäĻ░Ć ņ”ØĻ░ĆĒĢśņśĆĻ│Ā, Clostridium coccoidesņØś ĒÆŹļČĆļÅäļŖö Ļ░ÉņåīĒĢśņśĆļŗż. ņ£ä ņ▓┤ļČĆņŚÉņä£ļÅä LactobacillusņØś ĒÆŹļČĆļÅäļŖö ņ”ØĻ░ĆĒĢ£ ļ░śļ®┤ Clostridium coccoidesņØś ĒÆŹļČĆļÅäļŖö Ļ░ÉņåīĒĢśņśĆļŗż. ļŗżļ¦ī, ņØ┤ ņŚ░ĻĄ¼ļŖö ņżæĒĢ®ĒÜ©ņåī ņŚ░ņćäļ░śņØæļ▓ĢņØä ņØ┤ņÜ®ĒĢśņŚ¼ ļ¬®Ēæ£ļĪ£ ņé╝ņØĆ ņØ╝Ļ│▒ Ļ░Ćņ¦Ć ļ░ĢĒģīļ”¼ņĢä ĻĄ░ņŚÉ ļīĆĒĢ┤ņä£ļ¦ī ņŚ░ĻĄ¼ļź╝ ņ¦äĒ¢ēĒĢśņśĆĻĖ░ ļĢīļ¼ĖņŚÉ, ņŚ░ĻĄ¼ ļīĆņāüņØ┤ ņĢäļŗī ļŗżļźĖ ļ░ĢĒģīļ”¼ņĢäņØś ĒÆŹļČĆļÅäļéś ļ»ĖņāØļ¼╝ĻĄ░ņ¦æņØś ļŗżņ¢æņä▒ņØä ĒÖĢņØĖĒĢĀ ņłśļŖö ņŚåņŚłļŗż. ļśÉĒĢ£, ņŗŁņØ┤ņ¦ĆņןĻČżņ¢æ ĒÖśņ×Éļź╝ ļīĆņāüņ£╝ļĪ£ ĒĢśņśĆĻĖ░ ļĢīļ¼ĖņŚÉ ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ņĀ£ĻĘĀ ņ╣śļŻī ņØ┤Ēøä ņ¢æņä▒ņ×ÉĒÄīĒöäņ¢ĄņĀ£ņĀ£ļź╝ 2ņŻ╝Ļ░ä ņČöĻ░ĆļĪ£ Ēł¼ņĢĮĒĢśņśĆĻ│Ā, ļö░ļØ╝ņä£ ņ¢æņä▒ņ×ÉĒÄīĒöäņ¢ĄņĀ£ņĀ£ļĪ£ ņØĖĒĢ£ ņ£ä ļé┤ ļ»ĖņāØļ¼╝ĻĄ░ņ¦æ ņĪ░ņä▒ ļ│ĆĒÖö Ļ░ĆļŖźņä▒ņØä ņÖäņĀäĒ׳ ļ░░ņĀ£ĒĢĀ ņłś ņŚåņŚłļŗżļŖö ĒĢ£Ļ│äĻ░Ć ņ׳ļŗż.
2017ļģä Li ļō▒[11]ņØĆ ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ņĀ£ĻĘĀ ņ╣śļŻī ņĀä/Ēøä ņ£ä ļé┤ ļ»ĖņāØļ¼╝ĻĄ░ņ¦æņØś ņĪ░ņä▒ ļ│ĆĒÖöļź╝ ņĄ£ņ┤łļĪ£ 16S rRNA ņ£ĀņĀäņ×É ņŗ£ĒĆĆņŗ▒ņØä ĒåĄĒĢ┤ ļČäņäØĒĢśņśĆļŗż. ņ£äņŚ╝ ĒÖśņ×É 7ļ¬ģ, ņןņāüĒö╝ĒÖöņāØ ĒÖśņ×É 4ļ¬ģņŚÉ ļīĆĒĢ┤ ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ņĀ£ĻĘĀ ņ╣śļŻīļź╝ ņŗ£Ē¢ēĒĢ£ Ļ▓░Ļ│╝ ļ»ĖņāØļ¼╝ĻĄ░ņ¦æņØś ļŗżņ¢æņä▒ņØ┤ ņ”ØĻ░ĆĒĢśņśĆļŗż. ĒÆŹļČĆļÅäĻ░Ć ļŖśņ¢┤ļé£ ļ░ĢĒģīļ”¼ņĢä ļČäļźśĻĄ░ņØĆ ļīĆļČĆļČä non-Helicobacter ProteobacteriaņśĆļŖöļŹ░, Neisseria, Serratia, Haemophilus parainfluenzae, Rhizobium ļō▒ņØ┤ ņ׳ņŚłĻ│Ā, ņØ┤ ņÖĖņŚÉ Flavobacterium, Fusobacterium ļō▒ļÅä ņĀ£ĻĘĀ ņ╣śļŻī Ēøä ĒÆŹļČĆļÅäĻ░Ć ņ”ØĻ░ĆĒĢśņśĆļŗż. ņØ┤ ņŚ░ĻĄ¼ļŖö ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ Ļ░ÉņŚ╝ņ£╝ļĪ£ ņØĖĒĢ┤ Ļ░ÉņåīļÉ£ ļŗżņ¢æņä▒ņØ┤ ņĀ£ĻĘĀ ņ╣śļŻī Ēøä ļ│ĄņøÉļÉĀ ņłś ņ׳ņØīņØä ļ│┤ņŚ¼ņżĆ ņ▓½ ļ▓łņ¦Ė ņŚ░ĻĄ¼ņØ┤ĻĖ░ļŖö ĒĢśļéś, ņĀ£ĻĘĀ ņ╣śļŻī ļīĆņāüĻĄ░ņŚÉ ļŗ©ņł£ ņ£äņŚ╝ ĒÖśņ×ÉņÖĆ ņןņāüĒö╝ĒÖöņāØ ĒÖśņ×ÉĻ░Ć ņä×ņŚ¼ ņ׳ņ¢┤ ņĀ£ĻĘĀ ņ╣śļŻīņØś ĒÜ©Ļ│╝ļź╝ ņś©ņĀäĒ׳ ņØ┤ĒĢ┤ĒĢśĻĖ░ņŚÉļŖö ņĀ£ņĢĮņØ┤ ņ׳ņØä ņłś ņ׳ļŗż.
2019ļģä He ļō▒[12]Ļ│╝ Park ļō▒[13]ņØĆ ņ£äņČĢņä▒ ņ£äņŚ╝ ļ░Å ņןņāüĒö╝ĒÖöņāØņØ┤ ļ░£ņāØĒĢśņ¦Ć ņĢŖņØĆ ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ņŚ░Ļ┤Ć ņ£äņŚ╝ ĒÖśņ×ÉņŚÉ ļīĆĒĢ┤ņä£ ņĀ£ĻĘĀ ņ╣śļŻī ņĀä/Ēøä ņ£ä ļé┤ ļ»ĖņāØļ¼╝ĻĄ░ņ¦æņØś ļ│ĆĒÖöļź╝ ļČäņäØĒĢśņśĆļŗż. He ļō▒[12]ņØś ņŚ░ĻĄ¼ņŚÉņä£ļŖö ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ņĀ£ĻĘĀ ņ╣śļŻī ņĀäņŚÉ ļ╣äĒĢ┤ ņĀ£ĻĘĀ ņ╣śļŻī Ēøä ņ£ä ļé┤ ļ»ĖņāØļ¼╝ĻĄ░ņ¦æņØś ļŗżņ¢æņä▒ņØ┤ ņ”ØĻ░ĆĒĢ©ņØä ļ│┤ņśĆĻ│Ā, ņĀ£ĻĘĀ ņ╣śļŻī Ēøä 26ņŻ╝ Ļ▓ĮĻ│╝ ņŗ£ ņĀ£ĻĘĀ ņ╣śļŻī ņĀäņŚÉ ļ╣äĒĢ┤ Prevotella, Ralstonia, Pseudomonas, LactobacillusņØś ĒÆŹļČĆļÅäĻ░Ć ņ”ØĻ░ĆĒĢ©ņØä ļ│┤ņśĆļŗż. ņĀ£ĻĘĀ ņ╣śļŻī Ēøä 4ņŻ╝ņÖĆ 24ņŻ╝ Ļ▓ĮĻ│╝ĒĢ£ ļÆż Ļ░üĻ░ü ņ£ä ļé┤ ļ»ĖņāØļ¼╝ĻĄ░ņ¦æņØä ļČäņäØĒĢśņśĆļŖöļŹ░, RalstoniaļŖö 24ņŻ╝ņ¦ĖņŚÉ ļ╣äĒĢ┤ 4ņŻ╝ņ¦ĖņŚÉ ĒÆŹļČĆļÅäĻ░Ć ļåÆņĢśĻ│Ā, PseudomonasļŖö 4ņŻ╝ņ¦ĖņÖĆ 24ņŻ╝ņ¦ĖņŚÉ ĒÆŹļČĆļÅä ņ░©ņØ┤Ļ░Ć ņŚåņŚłĻ│Ā, Prevotella, LactobacillusļŖö 4ņŻ╝ņ¦ĖņŚÉ ļ╣äĒĢ┤ 24ņŻ╝ņ¦ĖņŚÉ ĒÆŹļČĆļÅäĻ░Ć ļŹö ļåÆņĢä, ņĀ£ĻĘĀ ņ╣śļŻī Ēøä ļŖśņ¢┤ļéśļŖö ļ░ĢĒģīļ”¼ņĢä ļČäļźśĻĄ░ļÅä ņŗ£ĻĖ░ņŚÉ ļö░ļØ╝ ļŗ¼ļØ╝ņ¦ł ņłś ņ׳ņØīņØä ļ│┤Ļ│ĀĒĢśņśĆļŗż. Park ļō▒[13]ņØĆ ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ņĀ£ĻĘĀ ņ╣śļŻī Ēøä Rhizobiales, Neisseriaceae ļō▒ņØś ĒÆŹļČĆļÅäĻ░Ć ņ”ØĻ░ĆĒĢ©ņØä ļ│┤ņśĆļŗż. ļŗżļźĖ ņŚ░ĻĄ¼ņÖĆ ļ¦łņ░¼Ļ░Ćņ¦ĆļĪ£ ņĀ£ĻĘĀ ņ╣śļŻī ņĀäņŚÉ ļ╣äĒĢ┤ ņĀ£ĻĘĀ ņ╣śļŻī Ēøä ļ»ĖņāØļ¼╝ĻĄ░ņ¦æņØś ņĪ░ņä▒ņØ┤ Ēü¼Ļ▓ī ļ│ĆĒÖöĒĢśņśĆņ£╝ļéś, ņĀ£ĻĘĀ ņ╣śļŻī Ēøä ļ»ĖņāØļ¼╝ĻĄ░ņ¦æņØś ņĪ░ņä▒ņØ┤ Firmicutes ļ░Å CyanobacteriaņØś ĒÆŹļČĆļÅäĻ░Ć ņāüļīĆņĀüņ£╝ļĪ£ ļåÆņØĆ Ļ▒┤Ļ░ĢņØĖņØś ļ»ĖņāØļ¼╝ĻĄ░ņ¦æļ│┤ļŗżļŖö Proteobacteria ļ░Å BacteroidetesņØś ĒÆŹļČĆļÅäĻ░Ć ņāüļīĆņĀüņ£╝ļĪ£ ļåÆņØĆ ņןņāüĒö╝ĒÖöņāØ ĒÖśņ×ÉņØś ļ»ĖņāØļ¼╝ĻĄ░ņ¦æņŚÉ Ļ░ĆĻ╣ØĻ▓ī ļ│ĆĒÖöĒĢ©ņØä ĒÖĢņØĖĒĢśņśĆļŗż[13]. ņØ┤ļŖö ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ Ļ░ÉņŚ╝ņØä ņ╣śļŻīĒĢśļŹöļØ╝ļÅä ņ£ä ļé┤ ļ»ĖņāØļ¼╝ĻĄ░ņ¦æņØś ņĪ░ņä▒ņØ┤ ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ Ļ░ÉņŚ╝ ņĀä ņāüĒā£ļĪ£ ņÖäņĀäĒ׳ ļ│ĄņøÉļÉśņ¦ĆļŖö ņĢŖņØä ņłśļÅä ņ׳ņØīņØä ņŗ£ņé¼ĒĢśļ®░, ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ņĀ£ĻĘĀ ņ╣śļŻīļ¦īņ£╝ļĪ£ ņ£äņĢöņØä 100% ņśłļ░®ĒĢĀ ņłś ņŚåļŖö ņØ┤ņ£ĀĻ░Ć ļÉĀ ņłśļÅä ņ׳ļŗż.
2019ļģä Serrano ļō▒[14]ņØś ņŚ░ĻĄ¼ņÖĆ 2020ļģä Miao ļō▒[16]ņØś ņŚ░ĻĄ¼ļŖö ņåīņĢä ĒÖśņ×ÉņŚÉņä£ņØś ņĀ£ĻĘĀ ņ╣śļŻī Ēøä ņ£ä ļé┤ ļ»ĖņāØļ¼╝ĻĄ░ņ¦æ ņĪ░ņä▒ ļ│ĆĒÖöļź╝ ļČäņäØĒĢśņśĆļŗż. ņåīņĢäņŚÉņä£ņØś ņŚ░ĻĄ¼ļŖö ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ļ¦īņä▒ Ļ░ÉņŚ╝ņØś ņ£Āļ│æ ĻĖ░Ļ░äņØ┤ ĻĖĖņ¦Ć ņĢŖņØä Ļ▓ĮņÜ░ ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ņĀ£ĻĘĀ ņ╣śļŻīņŚÉ ļö░ļźĖ ņ£äņĢö ļ░£ņāØ ņśłļ░® ĒÜ©Ļ│╝Ļ░Ć Ēü┤ ņłś ņ׳ļŗżļŖö Ļ░ĆņĀĢĒĢśņŚÉ, ņ£äņĢö ņśłļ░®ņŚÉ ļÅäņøĆņØ┤ ļÉśļŖö ņ£ä ļé┤ ļ»ĖņāØļ¼╝ĻĄ░ņ¦æņØś ĒŖ╣ņä▒ņØä ĒīīņĢģĒĢśļŖöļŹ░ ļÅäņøĆņØä ņżä ņłś ņ׳ļŗż. Serrano ļō▒[14]ņØĆ ņśżņŗ¼Ļ│╝ ļ│ĄļČĆļČłĒÄĖĻ░ÉņØä ĒśĖņåīĒĢśļŖö 7ļ¬ģņØś 13ņäĖ ņØ┤ĒĢś ņåīņĢä ĒÖśņ×Éļź╝ ļīĆņāüņ£╝ļĪ£ ņĀ£ĻĘĀ ņ╣śļŻīļź╝ ņŗ£Ē¢ēĒĢśņŚ¼, ņĀ£ĻĘĀ ņ╣śļŻī ņĀäņŚÉ ļ╣äĒĢ┤ ņĀ£ĻĘĀ ņ╣śļŻī Ēøä ņ£ä ļé┤ ļ»ĖņāØļ¼╝ĻĄ░ņ¦æņØś ļŗżņ¢æņä▒ņØ┤ ņ”ØĻ░ĆĒĢśĻ│Ā, ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ Ļ░ÉņŚ╝ņØ┤ ņŚåļŖö ņåīņĢäņÖĆ ņĀ£ĻĘĀ ņ╣śļŻīļź╝ ļ░øņØĆ ņåīņĢä Ļ░äņŚÉ ļ»ĖņāØļ¼╝ĻĄ░ņ¦æņØś ļŗżņ¢æņä▒ņŚÉ ņ░©ņØ┤Ļ░Ć ņŚåņØīņØä ļ│┤ņśĆļŗż. ņĀ£ĻĘĀ ņ╣śļŻī ĒøäņØś ļ»ĖņāØļ¼╝ĻĄ░ņ¦æņŚÉļŖö Pseudomonadaceae, Delftia, Kocuria, Stenotrophomonas ļō▒ņØś ĒÆŹļČĆļÅäĻ░Ć ņ”ØĻ░ĆĒĢśņśĆļŗż. Miao ļō▒[16]ņØś ņŚ░ĻĄ¼ņŚÉņä£ļŖö ĒÅēĻĘĀ 11ņäĖņØś ņåīņĢä ĒÖśņ×É 11ļ¬ģņŚÉ ļīĆĒĢ┤ ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ņĀ£ĻĘĀ ņ╣śļŻīļź╝ ņŗ£Ē¢ēĒĢśņśĆĻ│Ā, ņĀ£ĻĘĀ ņ╣śļŻī ņĀäņŚÉ ļ╣äĒĢ┤ ņĀ£ĻĘĀ ņ╣śļŻī Ēøä ļ»ĖņāØļ¼╝ĻĄ░ņ¦æņØś ļŗżņ¢æņä▒ņØ┤ ņ”ØĻ░ĆĒĢśņśĆņ£╝ļ®░ Neisseria, Curvibacter, HaemophilusņØś ĒÆŹļČĆļÅäĻ░Ć ņ”ØĻ░ĆĒĢśņśĆļŗż. ļśÉĒĢ£, ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ Ļ░ÉņŚ╝ņØ┤ ņŚåļŖö ņåīņĢäņÖĆ ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ņĀ£ĻĘĀ ņ╣śļŻīļź╝ ļ░øņØĆ ņåīņĢäņŚÉņä£ ļŗżņ¢æņä▒ ņ¦ĆĒæ£ņØĖ Shannon indexņÖĆ Simpson indexĻ░Ć ņ£Āņé¼ĒĢ©ņØä ļ│┤ņśĆļŗż. ļŗżļ¦ī, richnessļź╝ ļéśĒāĆļé┤ļŖö Chao1Ļ│╝ abundance-based coverage estimatorļŖö ņĀ£ĻĘĀ ņ╣śļŻī ņĀäņŚÉ ļ╣äĒĢ┤ ņĀ£ĻĘĀ ņ╣śļŻī Ēøä ņāüņŖ╣ĒĢśĻĖ░ļŖö ĒĢśņśĆņ£╝ļéś, Ļ▒┤Ļ░ĢĒĢ£ ņåīņĢäņŚÉ ļ╣äĒĢ┤ņä£ļŖö ņ£ĀņØśĒĢśĻ▓ī ļé«ņĢä ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ Ļ░ÉņŚ╝ ņĀäņØś richnessņŚÉ ļÅäļŗ¼ĒĢśņ¦ĆļŖö ļ¬╗ĒĢśņśĆļŗż. ļŗżļ¦ī, ņØ┤ ņŚ░ĻĄ¼ņŚÉņä£ ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ņĀ£ĻĘĀ ņ╣śļŻīļź╝ ļ░øņØĆ ĒÖśņ×ÉņØś ļŗżņłśļŖö ņŗŁņØ┤ņ¦ĆņןĻČżņ¢æņØ┤ ņ׳ņŚłļŖöļŹ░, ņŗŁņØ┤ņ¦ĆņןĻČżņ¢æ ĒÖśņ×ÉņŚÉņä£ ņ£äņĢö ņ£äĒŚśņØ┤ Ļ░ÉņåīĒĢ£ļŗżļŖö ņĀÉņØä Ļ░ÉņĢłĒĢĀ ļĢī, ļ│Ė ņŚ░ĻĄ¼ Ļ▓░Ļ│╝ļź╝ ĒåĀļīĆļĪ£ ņ£äņĢö ļ░£ņāØĻ│╝ ņŚ░Ļ┤Ć ņ׳ļŖö ļ░ĢĒģīļ”¼ņĢäļź╝ ņČöļĪĀĒĢśĻĖ░ņŚÉļŖö ĒĢ£Ļ│äĻ░Ć ņ׳ļŗż[20].
2020ļģä Guo ļō▒[15]ņØĆ ņ┤Ø 58ļ¬ģņØś ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ Ļ░ÉņŚ╝ ĒÖśņ×ÉņŚÉ ļīĆĒĢ┤ ņĀ£ĻĘĀ ņ╣śļŻīļź╝ ņŗ£Ē¢ēĒĢśņśĆņ£╝ļ®░, ņĀ£ĻĘĀ ņ╣śļŻī ņä▒Ļ│Ą ĒÖśņ×ÉņØś ļ»ĖņāØļ¼╝ĻĄ░ņ¦æņØä ņĀ£ĻĘĀ ņ╣śļŻī ņŗżĒī© ĒÖśņ×ÉņÖĆ ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ļ»ĖĻ░ÉņŚ╝ņ×ÉņØś ļ»ĖņāØļ¼╝ĻĄ░ņ¦æĻ│╝ ļ╣äĻĄÉĒĢśņśĆļŗż. ņØ┤ņĀäņØś ļŗżļźĖ ņŚ░ĻĄ¼ņÖĆ ļ¦łņ░¼Ļ░Ćņ¦ĆļĪ£, ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ Ļ░ÉņŚ╝ ĒÖśņ×ÉņŚÉņä£ ļé«ņĢäņĀĖ ņ׳ļŖö ļ»ĖņāØļ¼╝ĻĄ░ņ¦æņØś ļŗżņ¢æņä▒ņØ┤, ņĀ£ĻĘĀ ņ╣śļŻī ņä▒Ļ│Ą ņŗ£ ņ”ØĻ░ĆĒĢśļŖö Ļ▓āņØä ĒÖĢņØĖĒĢśņśĆļŗż. ņĀ£ĻĘĀ ņ╣śļŻī ņä▒Ļ│Ą ņŗ£ ļŗżņ¢æņä▒ņØĆ ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ļ»ĖĻ░ÉņŚ╝ņ×ÉņØś ņ£ä ļé┤ ļ»ĖņāØļ¼╝ĻĄ░ņ¦æĻ│╝ ļ╣äņŖĘĒĢ£ ņłśņżĆņ£╝ļĪ£ ļ│ĄņøÉļÉśņŚłņ£╝ļ®░, ņĀ£ĻĘĀ ņ╣śļŻī ņŗżĒī© ņŗ£ņŚÉļŖö ņĀ£ĻĘĀ ņ╣śļŻīļź╝ ņŗ£Ē¢ēļ░øĻĖ░ ņĀäĻ│╝ ļ╣äņŖĘĒĢ£ ņĀĢļÅäņØś ļŗżņ¢æņä▒ņØä ļ│┤ņśĆļŗż. ņĀ£ĻĘĀ ņ╣śļŻī ņä▒Ļ│Ą ņŗ£ ņ£ä ļé┤ ļ»ĖņāØļ¼╝ĻĄ░ņ¦æņŚÉļŖö Staphylococcus, Corynebacterium, Fusobacterium, Bacteroides, Streptophyta, Prevotella, Gemella, Porphyromonas, Alloprevotella, Veillonella, Neisseria, Streptococcus, Rothia, HaemophilusņØś ĒÆŹļČĆļÅäĻ░Ć ņ”ØĻ░ĆĒĢśņśĆļŗż. ĻĘĖļ¤¼ļéś ņŻ╝ņä▒ļČä ļČäņäØ(principle component analysis)ņØä ĒåĄĒĢ┤ ņĀäņ▓┤ņĀüņØĖ ļ»ĖņāØļ¼╝ĻĄ░ņ¦æ ņĪ░ņä▒ņØä ļ╣äĻĄÉĒĢśņśĆņØä ļĢīļŖö ņĀ£ĻĘĀ ņ╣śļŻī ņä▒Ļ│Ą ĒÖśņ×ÉņØś ļ»ĖņāØļ¼╝ĻĄ░ņ¦æņØ┤ ņĀ£ĻĘĀ ņ╣śļŻī ņĀä ļ»ĖņāØļ¼╝ĻĄ░ņ¦æĻ│╝ ņ░©ņØ┤Ļ░Ć ņ׳ņŚłņ¦Ćļ¦ī, ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ļ»ĖĻ░ÉņŚ╝ņ×ÉņØś ļ»ĖņāØļ¼╝ĻĄ░ņ¦æĻ│╝ ļ╣äĻĄÉĒĢ┤ļÅä ņ░©ņØ┤Ļ░Ć ņ׳ņŚłļŗż. ņĀĆņ×ÉļōżņØĆ ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ņĀ£ĻĘĀ ņ╣śļŻī Ēøä ņ£ä ļé┤ ļ»ĖņāØļ¼╝ĻĄ░ņ¦æņØ┤ ļ│ĄņøÉļÉĀ ņłś ņ׳ņØīņØä ņŻ╝ļĪ£ Ļ░ĢņĪ░ĒĢśĻĖ░ļŖö ĒĢśņśĆņ£╝ļéś, ņĀ£ĻĘĀ ņ╣śļŻīļ¦īņ£╝ļĪ£ ņ£ä ļé┤ ļ»ĖņāØļ¼╝ĻĄ░ņ¦æņØ┤ ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ Ļ░ÉņŚ╝ ņĀäĻ│╝ Ļ░ÖņØ┤ ņÖäņĀäĒ׳ ņĀĢņāüĒÖö ļÉśļŖöĻ░ĆņŚÉ ļīĆĒĢ┤ņä£ļŖö ņØśļ¼ĖņØ┤ ļé©ļŖöļŗż.
Guo ļō▒[15]ņØś ņŚ░ĻĄ¼ņŚÉņä£ ļ│┤ņŚ¼ņżĆ ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ņĀ£ĻĘĀ ņ╣śļŻīņŚÉ ļö░ļźĖ ļ»ĖņāØļ¼╝ĻĄ░ņ¦æ ļŗżņ¢æņä▒ņØś ļ│ĄņøÉņØĆ ļ╣äĻĄÉĻĄ░ņØĖ ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ļ»ĖĻ░ÉņŚ╝ņ×ÉĻĄ░ņŚÉ ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ Ļ░ÉņŚ╝ņØ┤ ņŚåņŚłļŹś Ļ▒┤Ļ░ĢņØĖĻ│╝ ņ£äņČĢņä▒ ņ£äņŚ╝ ļ░Å ņןņāüĒö╝ĒÖöņāØņØ┤ ņ¦äĒ¢ēļÉśņ¢┤ ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ Ļ░ÉņŚ╝ņØ┤ ņåīņŗżļÉ£ Ļ│╝Ļ▒░ Ļ░ÉņŚ╝ņ×ÉĻ░Ć ĒĢ©Ļ╗ś ĒżĒĢ©ļÉśņŚłĻĖ░ ļĢīļ¼ĖņØ╝ Ļ░ĆļŖźņä▒ņØ┤ ņ׳ļŗż. ņØ┤ņŚÉ, ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ļ»ĖĻ░ÉņŚ╝ņ×Éļź╝ Ļ▒┤Ļ░ĢņØĖĻ│╝ ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ Ļ│╝Ļ▒░ Ļ░ÉņŚ╝ņ×ÉļĪ£ ļČäļ”¼ĒĢśņŚ¼ ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ņĀ£ĻĘĀ ņ╣śļŻī ĒÖśņ×ÉņÖĆ ļ╣äĻĄÉĒĢśĻĖ░ ņ£äĒĢ┤, Guo ļō▒[15]ņØ┤ ņŗ£Ē¢ēĒĢ£ ļČäņäØņØä Park ļō▒[13]ņØś ņŚ░ĻĄ¼ ļŹ░ņØ┤Ēä░ļź╝ ņØ┤ņÜ®ĒĢ┤ ņ×¼ĒśäĒĢ┤ ļ│┤ņĢśļŗż(Fig. 1, 2). Guo ļō▒[15]ņØś ņŚ░ĻĄ¼ Ļ▓░Ļ│╝ņÖĆ ļ¦łņ░¼Ļ░Ćņ¦ĆļĪ£ ņĀ£ĻĘĀ ņ╣śļŻī ņŗżĒī© ņŗ£ richness indexņÖĆ Shannon indexĻ░Ć ņĀ£ĻĘĀ ņ╣śļŻī ņĀäņØś ņłśņ╣śņÖĆ ļ╣äņŖĘĒĢ£ ņłśņżĆņŚÉ ļ©Ėļ¼╝ļ¤¼ ņ׳ļŖö Ļ▓āņŚÉ ļ╣äĒĢ┤, ņĀ£ĻĘĀ ņ╣śļŻī ņä▒Ļ│Ą ņŗ£ņŚÉļŖö ņłśņ╣śĻ░Ć ņāüņŖ╣ĒĢ©ņØä ĒÖĢņØĖĒĢśņśĆļŗż(Fig. 1A, B). ĻĘĖļ¤¼ļéś ņāüņŖ╣ĒĢ£ richness indexņÖĆ Shannon indexļŖö ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ Ļ│╝Ļ▒░ Ļ░ÉņŚ╝ņ×ÉņÖĆ ļ╣äņŖĘĒĢ£ ņłśņżĆĻ╣īņ¦Ćļ¦ī ņāüņŖ╣ĒĢśņśĆĻ│Ā, Ļ▒┤Ļ░ĢņØĖņØś ņłśņżĆņŚÉļŖö ļÅäļŗ¼ĒĢśņ¦Ć ļ¬╗ĒĢśņśĆļŗż. Guo ļō▒[15]ņØĆ ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ņĀ£ĻĘĀ ņ╣śļŻī ņä▒Ļ│Ą ņŗ£ ĒÆŹļČĆļÅäĻ░Ć ņ”ØĻ░ĆĒĢśļŖö 17Ļ░£ņØś generaņÖĆ ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ņØś ĒÆŹļČĆļÅäļź╝ ņØ┤ņÜ®ĒĢśņŚ¼ ļ»ĖņāØļ¼╝ ļČłĻĘĀĒśĢ ņ¦ĆĒæ£(microbial dysbiosis index)ļź╝ ņĀ£ņŗ£ĒĢśņśĆļŖöļŹ░, ņØ┤ ņ¦ĆĒæ£ ņŚŁņŗ£ ņĀ£ĻĘĀ ņ╣śļŻī ņä▒Ļ│Ą ņŗ£ Ļ▒┤Ļ░ĢņØĖļ│┤ļŗżļŖö ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ Ļ│╝Ļ▒░ Ļ░ÉņŚ╝ņ×ÉņÖĆ ņ£Āņé¼ĒĢśĻ▓ī ļ│ĆĒÖöĒĢśņśĆļŗż(Fig. 1C). ļśÉĒĢ£, ņŻ╝ņä▒ļČä ļČäņäØ Ļ▓░Ļ│╝ņŚÉņä£ļÅä ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ņĀ£ĻĘĀ ņ╣śļŻī ņä▒Ļ│Ą ņŗ£ ņ£ä ļé┤ ļ»ĖņāØļ¼╝ĻĄ░ņ¦æņØś ņĪ░ņä▒ņØĆ Ļ▒┤Ļ░ĢņØĖļ│┤ļŗżļŖö ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ Ļ│╝Ļ▒░ Ļ░ÉņŚ╝ņ×ÉņÖĆ ļ╣äņŖĘĒĢśņśĆļŗż(Fig. 2).
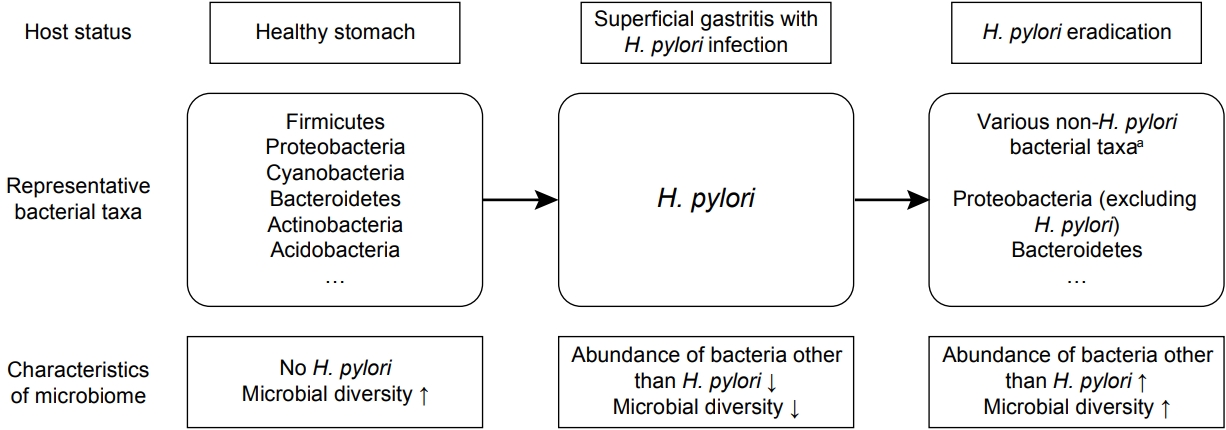
ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ņĀ£ĻĘĀ ņ╣śļŻī Ēøä ņ£ä ļé┤ ļ»ĖņāØļ¼╝ĻĄ░ņ¦æņØś ļ│ĆĒÖöļź╝ ļČäņäØĒĢ£ Ēśäņ×¼Ļ╣īņ¦ĆņØś ļ¬©ļōĀ ņŚ░ĻĄ¼Ļ░Ć ņØ╝Ļ┤ĆļÉśĻ▓ī ļ│┤ņŚ¼ņŻ╝ļŖö ņåīĻ▓¼ņØĆ ņĀ£ĻĘĀ ņ╣śļŻī ņä▒Ļ│Ą ņŗ£ ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ņÖĖ ļŗżļźĖ ņŚ¼ļ¤¼ ļ░ĢĒģīļ”¼ņĢäņØś ĒÆŹļČĆļÅäĻ░Ć ņ”ØĻ░ĆĒĢśĻ│Ā ļ»ĖņāØļ¼╝ĻĄ░ņ¦æņØś ļŗżņ¢æņä▒ņØ┤ ņ”ØĻ░ĆĒĢ£ļŗżļŖö ņĀÉņØ┤ļŗż(Fig. 3). ĻĘĖļ¤¼ļéś ņĀ£ĻĘĀ ņ╣śļŻī Ēøä ņ¢┤ļ¢ż ļ░ĢĒģīļ”¼ņĢä ļČäļźśĻĄ░ņØ┤ ņāüļīĆņĀüņ£╝ļĪ£ ļåÆņØĆ ļ╣äņ£©ļĪ£ ņ”ØĻ░ĆĒĢśļŖöņ¦ĆļŖö ņŚ░ĻĄ¼ļ¦łļŗż ņ░©ņØ┤Ļ░Ć ņ׳Ļ│Ā, ņĀ£ĻĘĀ ņ╣śļŻī Ēøä ļ»ĖņāØļ¼╝ĻĄ░ņ¦æņØś ņĪ░ņä▒Ļ│╝ ļŗżņ¢æņä▒ņØ┤ ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ Ļ░ÉņŚ╝ ņØ┤ņĀäĻ│╝ Ļ░ÖņØ┤ ņÖäņĀäĒ׳ ļ│ĄņøÉļÉśļŖöĻ░ĆņŚÉ ļīĆĒĢ┤ņä£ļŖö ņŚ░ĻĄ¼ļ¦łļŗż ņ░©ņØ┤Ļ░Ć ņ׳ļŗż. ņØ┤ļŖö ņ£ä ļé┤ ļ»ĖņāØļ¼╝ĻĄ░ņ¦æņØś ņĪ░ņä▒ņŚÉ ņŚ¼ļ¤¼ ņ¦ĆņŚŁņĀü, ĒÖśĻ▓ĮņĀü ņØĖņ×ÉĻ░Ć Ļ┤ĆņŚ¼ĒĢ©ņØĆ ļ¼╝ļĪĀņØ┤Ļ│Ā, ņŚ░ĻĄ¼ ļīĆņāüņ×ÉņØś ņ£ä ļé┤ ņ¦łļ│æ ņāüĒā£(ņ£äņŚ╝, ņ£äņČĢņä▒ ņ£äņŚ╝, ņןņāüĒö╝ĒÖöņāØ)Ļ░Ć ņŚ░ĻĄ¼ļ¦łļŗż ņ░©ņØ┤Ļ░Ć ņ׳ĻĖ░ ļĢīļ¼Ėņ£╝ļĪ£ ņāØĻ░üļÉ£ļŗż[8,21]. Ļ░Ćņן ņĄ£ĻĘ╝ņŚÉ ļ░£Ēæ£ļÉ£ 2020ļģä Sung ļō▒[6]ņØś ņŚ░ĻĄ¼ļŖö 102ļ¬ģņØś ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ Ļ░ÉņŚ╝ņ×ÉņŚÉĻ▓ī ņĀ£ĻĘĀ ņ╣śļŻīļź╝ ņŗ£Ē¢ēĒĢśĻ│Ā 1ļģä Ēøä ņ£äņŚ╝ņØ┤ ņ¦ĆņåŹļÉśĻ▒░ļéś, ņ£äņČĢņä▒ ņ£äņŚ╝ Ēś╣ņØĆ ņןņāüĒö╝ĒÖöņāØņØ┤ ņāłļĪ£ ļ░£ņāØĒĢ£ ĒÖśņ×ÉņØś ļ»ĖņāØļ¼╝ĻĄ░ņ¦æņØś ĒŖ╣ņä▒ņØä ļČäņäØĒĢśņśĆļŗż. ĒØźļ»ĖļĪŁĻ▓īļÅä, ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ņĀ£ĻĘĀ ņ╣śļŻīĻ░Ć ņä▒Ļ│ĄĒĢśņśĆļŹöļØ╝ļÅä ļ»ĖņāØļ¼╝ĻĄ░ņ¦æņØś ņĪ░ņä▒ņØĆ ĒÖśņ×É Ļ░äņŚÉ ņ░©ņØ┤Ļ░Ć ņ׳ņŚłļŖöļŹ░, 1ļģä Ēøä ņ£äņŚ╝ņØ┤ ņ¦ĆņåŹļÉśņŚłļŹś ĒÖśņ×ÉņŚÉņä£ļŖö ĻĘĖļĀćņ¦Ć ņĢŖņØĆ ĒÖśņ×Éļ│┤ļŗż Acinetobacter lwoffii, Streptococcus anginosus, RalstoniaņØś ĒÆŹļČĆļÅäĻ░Ć ņ”ØĻ░ĆĒĢśņśĆĻ│Ā, ņ£äņČĢņä▒ ņ£äņŚ╝ņØ┤ ņāłļĪ£ ļ░£ņāØĒĢ£ ĒÖśņ×ÉņŚÉņä£ļŖö ĻĘĖļĀćņ¦Ć ņĢŖņØĆ ĒÖśņ×Éļ│┤ļŗż Granulicatella, Streptococcus, Rothia, LeptotrichiaņØś ĒÆŹļČĆļÅäĻ░Ć ņ”ØĻ░ĆĒĢśņśĆņ£╝ļ®░, ņןņāüĒö╝ĒÖöņāØņØ┤ ņāłļĪ£ ļ░£ņāØĒĢ£ ĒÖśņ×ÉņŚÉņä£ļŖö ĻĘĖļĀćņ¦Ć ņĢŖņØĆ ĒÖśņ×Éļ│┤ļŗż Mesorhizobium, CupriavidusņØś ĒÆŹļČĆļÅäĻ░Ć ņ”ØĻ░ĆĒĢśņśĆļŗż. Sung ļō▒[6]ņØĆ ņØ┤ļōż ļ»ĖņāØļ¼╝ ļČäļźśĻĄ░ņØ┤ ņ×Āņ×¼ņĀüņ£╝ļĪ£ ņ£äņĢö ļ░£ļ│æņŚÉ ĻĖ░ņŚ¼ĒĢśļŖö ņ£ä ļé┤ ļ»ĖņāØļ¼╝ņØ┤Ļ│Ā ņ£äņĢö ņśłļ░®ņØś ĒāĆĻ▓¤ņØ┤ ļÉĀ ņłśļÅä ņ׳ņØīņØä Ļ░ĢņĪ░ĒĢśņśĆļŗż. ļ¼╝ļĪĀ, ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ņĀ£ĻĘĀ ņ╣śļŻī 1ļģä Ēøä ņןņāüĒö╝ĒÖöņāØņØ┤ ņāłļĪ£ņØ┤ ļ░£ņāØĒĢ£ ĒÖśņ×ÉņŚÉņä£ ļ│┤ļŗż ļ¦ÄņØ┤ ļ░£Ļ▓¼ļÉśļŖö Mesorhizobium Ēś╣ņØĆ CupriavidusņÖĆ Ļ░ÖņØĆ ļ░ĢĒģīļ”¼ņĢäĻ░Ć ņŗżņĀ£ļĪ£ ņןņāüĒö╝ĒÖöņāØņØä ņ£Āļ░£ĒĢ£ ļ»ĖņāØļ¼╝ņØĖņ¦Ć, ņĢäļŗłļ®┤ ņןņāüĒö╝ĒÖöņāØņØ┤ ļ░£ņāØĒĢśņśĆĻĖ░ ļĢīļ¼ĖņŚÉ ņØ┤ņ░©ņĀüņ£╝ļĪ£ ņ”ØņŗØļÉ£ ļ»ĖņāØļ¼╝ņØĖņ¦Ć ĒīÉļŗ©ĒĢśĻĖ░ļŖö ņĢäņ¦ü ņØ┤ļź┤ļéś, ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ņĀ£ĻĘĀ ņ╣śļŻīļź╝ ļ░øņØĆ ļ¬©ļōĀ ĒÖśņ×ÉņŚÉņä£ ņ£ä ļé┤ ļ»ĖņāØļ¼╝ĻĄ░ņ¦æņØ┤ Ļ▒┤Ļ░ĢņØĖĻ│╝ Ļ░ÖņØĆ ņłśņżĆņ£╝ļĪ£ ļ│ĄņøÉļÉśļŖö Ļ▓āņØĆ ņĢäļŗī Ļ▓āņ£╝ļĪ£ ņāØĻ░üļÉ£ļŗż.
ņ¦ĆĻĖłĻ╣īņ¦Ć ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ņĀ£ĻĘĀ ņ╣śļŻī Ēøä ņ£ä ļé┤ ļ»ĖņāØļ¼╝ĻĄ░ņ¦æņØś ļ│ĆĒÖöļź╝ ļ│Ė ņŚ░ĻĄ¼ļŖö ļīĆļČĆļČä 16S rRNA ņ£ĀņĀäņ×É ņŗ£ĒĆĆņŗ▒ņØä ņØ┤ņÜ®ĒĢśņŚ¼ ņ¦äĒ¢ēļÉśņŚłļŗż. 16S rRNA ņ£ĀņĀäņ×É ņŗ£ĒĆĆņŗ▒ņØä ņØ┤ņÜ®ĒĢ£ ļ»ĖņāØļ¼╝ĻĄ░ņ¦æ ņŚ░ĻĄ¼ļŖö ļ░ĢĒģīļ”¼ņĢäņØś 16S rRNA ņ£ĀņĀäņ×É ņŚ╝ĻĖ░ ņä£ņŚ┤ļ¦īņØä ļČäņäØĒĢśņŚ¼ ņ¢┤ļ¢ż ļČäļźśĻĄ░ņØś ļ░ĢĒģīļ”¼ņĢäĻ░Ć ņ¢┤ļŖÉ ņĀĢļÅä ņĪ┤ņ×¼ĒĢśļŖöĻ░Ćļź╝ ņĢīņĢäļé┤ļŖö Ļ▓āņØ┤ļ®░ ļ®öĒāĆņ£ĀņĀäņ▓┤ņØś ļ¬©ļōĀ ņ£ĀņĀäņ×É ņŚ╝ĻĖ░ ņä£ņŚ┤ņØä ņ¦üņĀæ ļČäņäØĒĢśļŖö Ļ▓āņØ┤ ņĢäļŗłĻĖ░ ļĢīļ¼ĖņŚÉ, ļ®öĒāĆņ£ĀņĀäņ▓┤ņØś ĻĖ░ļŖźņØä ņ¦üņĀæņĀüņ£╝ļĪ£ ĒīīņĢģĒĢĀ ņłśļŖö ņŚåļŗż. ĻĘĖļ¤¼ļéś ĒåĀņ¢æņØ┤ļéś ņŗØļ¼╝ ļō▒ņŚÉ ņĪ┤ņ×¼ĒĢśļŖö ļ®öĒāĆņ£ĀņĀäņ▓┤ņÖĆļŖö ļŗ¼ļ”¼ ņé¼ļ×īņØś ņן ļé┤ņŚÉ ņĪ┤ņ×¼ĒĢśļŖö ļ®öĒāĆņ£ĀņĀäņ▓┤ļŖö ļīĆļČĆļČä ĻĘĖ ĻĖ░ļŖźņØ┤ ņĢīļĀżņĀĖ ņ׳ĻĖ░ ļĢīļ¼ĖņŚÉ, ņé¼ļ×īņŚÉ ļīĆĒĢ£ ļ»ĖņāØļ¼╝ĻĄ░ņ¦æ ņŚ░ĻĄ¼ņØś Ļ▓ĮņÜ░ņŚÉļŖö 16S rRNA ņŗ£ĒĆĆņŗ▒ņØä ĒåĄĒĢ┤ ņĢīņĢäļéĖ operational taxonomic unitņŚÉ ļīĆĒĢ£ ņĀĢļ│┤ļ¦īņØä Ļ░¢Ļ│Ā ļ®öĒāĆņ£ĀņĀäņ▓┤ņØś ĻĖ░ļŖźņØä ļ╣äĻĄÉņĀü ņĀĢĒÖĢĒ׳ ņČöļĪĀĒĢĀ ņłś ņ׳ļŗż[22,23].
Table 1ņŚÉ ņĀ£ņŗ£ļÉ£ 8Ļ▒┤ņØś ņŚ░ĻĄ¼ ņżæ 5Ļ▒┤ņØś ņŚ░ĻĄ¼ņŚÉņä£ ļ®öĒāĆņ£ĀņĀäņ▓┤ ĻĖ░ļŖźņØä ļČäņäØĒĢśņśĆĻ│Ā, ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ņĀ£ĻĘĀ ņ╣śļŻīņŚÉ ļö░ļźĖ ļ®öĒāĆņ£ĀņĀäņ▓┤ņØś ĻĖ░ļŖź ļ│ĆĒÖöļŖö 4Ļ▒┤ņØś ņŚ░ĻĄ¼ņŚÉņä£ ļ│┤Ļ│ĀļÉśņŚłļŗż[6,12,13,15]. He ļō▒[12]ņØĆ ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ņĀ£ĻĘĀ ņ╣śļŻī ņĀäņŚÉ ļ╣äĒĢ┤ ņĀ£ĻĘĀ ņ╣śļŻī Ēøä lipopolysaccharide biosynthesis, lipopolysaccharide biosynthesis proteins, bacterial motility proteins, bacterial chemotaxis Ļ┤ĆļĀ© ĻĖ░ļŖźņØ┤ Ļ░ÉņåīĒĢśĻ│Ā, glycolysis/gluconeogenesis, glycine, serine, threonine metabolism, pentose phosphate pathway Ļ┤ĆļĀ© ĻĖ░ļŖźņØĆ ņ”ØĻ░ĆĒĢ©ņØä ļ│┤Ļ│ĀĒĢśņśĆļŗż. Park ļō▒[13]ņØś ņŚ░ĻĄ¼ņŚÉņä£ļŖö Ļ▒┤Ļ░ĢņØĖņŚÉ ļ╣äĒĢ┤ ņןņāüĒö╝ĒÖöņāØ ĒÖśņ×ÉņŚÉņä£ ņ”ØĻ░ĆĒĢ£ ļŗżņ¢æĒĢ£ ļ®öĒāĆņ£ĀņĀäņ▓┤ ņ£ĀņĀäņ×Éļź╝ ļ│┤Ļ│ĀĒĢśņśĆļŖöļŹ░ ņØ┤ ņżæ type IV secretion system (T4SS) ļŗ©ļ░▒ņ¦ł ņĮöļö® ņ£ĀņĀäņ×ÉĻ░Ć ņ”ØĻ░ĆĒĢ┤ ņ׳ņŚłĻ│Ā, ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ Ļ░ÉņŚ╝ ņ£äņŚ╝ ĒÖśņ×ÉņŚÉņä£ ņĀ£ĻĘĀ ņ╣śļŻīļź╝ ņŗ£Ē¢ēĒĢ£ ĒøäņŚÉļÅä ļ®öĒāĆņ£ĀņĀäņ▓┤ņŚÉ T4SS ļŗ©ļ░▒ņ¦ł ņĮöļö® ņ£ĀņĀäņ×ÉĻ░Ć ņ”ØĻ░ĆĒĢśņśĆņØīņØä ļ│┤Ļ│ĀĒĢśņśĆļŗż. T4SS ļŗ©ļ░▒ņ¦łņØĆ ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ņØś CagA ļŗ©ļ░▒ņ¦łņØ┤ ņé¼ļ×īņØś ņ£ä ņāüĒö╝ņäĖĒżļĪ£ ņĀäļŗ¼ļÉśļŖöļŹ░ ĒĢäņłśņĀüņØĖ ļŗ©ļ░▒ņ¦łļĪ£, ņןņāüĒö╝ĒÖöņāØ ĒÖśņ×ÉņØś ļ®öĒāĆņ£ĀņĀäņ▓┤ņŚÉņä£ ņ”ØĻ░ĆĒĢśļŖö T4SS ļŗ©ļ░▒ņ¦ł ņĮöļö® ņ£ĀņĀäņ×ÉļŖö ņ£äņĢöņØś ļ░£ļ│æ ņ£äĒŚś ņ”ØĻ░ĆņÖĆ Ļ┤ĆļĀ©ņØ┤ ņ׳ņØä Ļ░ĆļŖźņä▒ņØ┤ ņĀ£ņŗ£ļÉśņŚłļŗż[13]. ļśÉĒĢ£, ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ņĀ£ĻĘĀ ņ╣śļŻīļź╝ ņŗ£Ē¢ēĒĢĀ Ļ▓ĮņÜ░ ļ®öĒāĆņ£ĀņĀäņ▓┤ņŚÉ T4SS ļŗ©ļ░▒ņ¦ł ņĮöļö® ņ£ĀņĀäņ×ÉĻ░Ć ņ”ØĻ░ĆĒĢ©ņŚÉļÅä ļČłĻĄ¼ĒĢśĻ│Ā ņ£äņĢöņØś ļ░£ļ│æ ņ£äĒŚśņØ┤ Ļ░ÉņåīĒĢ£ļŗżļŖö ņĀÉņØĆ ņ£äņĢöņØś ļ░£ņāØņŚÉ ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ņÖĖ ļ░ĢĒģīļ”¼ņĢäņØś ņĪ┤ņ×¼ļÅä ņżæņÜöĒĢĀ ņłś ņ׳ņ¦Ćļ¦ī ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ņ×Éņ▓┤Ļ░Ć ļ│┤ļŗż ņżæņÜöĒĢ£ ņØĖņ×Éņ×äņØä ņŗ£ņé¼ĒĢ£ļŗż[13].
Guo ļō▒[15]ņØĆ ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ņĀ£ĻĘĀ ņ╣śļŻī Ēøä ļ®öĒāĆņ£ĀņĀäņ▓┤ņŚÉ ļŗ©ļ░▒ņ¦łņØś ņåīĒÖöņÖĆ ĒØĪņłś, glucosaminoglycanĻ│╝ ĻĖ░ĒāĆ glycanņØś ļČäĒĢ┤, ņ£äņé░ ļČäļ╣ä ļ░Å ĒāäņłśĒÖöļ¼╝ņØś ņåīĒÖöņÖĆ ĒØĪņłś Ļ┤ĆļĀ© ĻĖ░ļŖźņØ┤ ņ”ØĻ░ĆĒĢśĻ│Ā, ĒÄĖļ¬© ļČĆņåŹņןņ╣ś(flagellar assembly), ņäĖĻĘĀņØś ĒÖöĒĢÖņŻ╝ņä▒(chemotaxis) ļ░Å ņäĖĻĘĀļČäļ╣äņ▓┤Ļ│ä Ļ┤ĆļĀ© ĻĖ░ļŖźņØ┤ ņżäņ¢┤ļō”ņØä ļ│┤ņŚ¼ņŻ╝ņŚłļŗż. Sung ļō▒[6]ņØś ņŚ░ĻĄ¼ņŚÉņä£ļŖö ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ņĀ£ĻĘĀ ņ╣śļŻī 1ļģä Ļ▓ĮĻ│╝ Ēøä, ņןņāüĒö╝ĒÖöņāØņØ┤ ļ░£ņāØĒĢśņ¦Ć ņĢŖņØĆ ĒÖśņ×ÉņØś ļ®öĒāĆņ£ĀņĀäņ▓┤ņŚÉ ļ╣äĒĢ┤ ņןņāüĒö╝ĒÖöņāØ ņØ┤ ņāłļĪ£ ļ░£ņāØĒĢ£ ĒÖśņ×ÉņØś ļ®öĒāĆņ£ĀņĀäņ▓┤ņŚÉņä£ ņĢäļ»ĖļģĖņé░ ļ░Å inositol phosphate ļīĆņé¼ Ļ┤ĆļĀ© ĻĖ░ļŖź ņ£ĀņĀäņ×ÉņÖĆ T4SS ļŗ©ļ░▒ņ¦ł ņĮöļö® ņ£ĀņĀäņ×ÉĻ░Ć ņ”ØĻ░ĆĒĢ©ņØä ĒÖĢņØĖĒĢśņśĆļŗż.
ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ņĀ£ĻĘĀ ņ╣śļŻī Ēøä ļŗżņ¢æĒĢ£ ļ░ĢĒģīļ”¼ņĢä ļČäļźśĻĄ░ņØś ĒÆŹļČĆļÅäĻ░Ć ņ”ØĻ░ĆĒĢśĻ│Ā, ĻĘĖņŚÉ ļö░ļØ╝ ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ņĀ£ĻĘĀ ņ╣śļŻī Ēøä ļ®öĒāĆņ£ĀņĀäņ▓┤ņØś ĻĖ░ļŖź ņŚŁņŗ£ ļ¦ÄņØĆ ļ│ĆĒÖöļź╝ ļ│┤ņ×äņØ┤ ņŚ¼ļ¤¼ ņŚ░ĻĄ¼ņŚÉņä£ ĒÖĢņØĖļÉśņŚłļŗż. ĻĘĖļ¤¼ļéś ņĢ×ņä£ ņé┤ĒÄ┤ļ│Ė ļ░öņÖĆ Ļ░ÖņØ┤ ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ņĀ£ĻĘĀ ņ╣śļŻī Ēøä ņ”ØĻ░ĆĒĢśļŖö ļ░ĢĒģīļ”¼ņĢä ļČäļźśĻĄ░ņØĆ ņŚ░ĻĄ¼ļ¦łļŗż ņ░©ņØ┤ļź╝ ļ│┤ņØ┤ļ®░, ļ®öĒāĆņ£ĀņĀäņ▓┤ņØś ĻĖ░ļŖź ļ│ĆĒÖö ņŚŁņŗ£ ņŚ░ĻĄ¼ņŚÉ ļö░ļØ╝ ņ░©ņØ┤Ļ░Ć ņ׳ļŗż. ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ņĀ£ĻĘĀ ņ╣śļŻī Ēøä ļ│ĆĒÖöĒĢśļŖö ļ®öĒāĆņ£ĀņĀäņ▓┤ņØś ĻĖ░ļŖźņØä ĒāÉņāēĒĢśļŖö Ļ▓āņØĆ ņ£äņĢöņØś ļ░£ļ│æ ĻĖ░ņĀä ĻĘ£ļ¬ģņŚÉ ļÅäņøĆņØä ņżä ņłśļŖö ņ׳ņ£╝ļéś, ņĢäņ¦üĻ╣īņ¦Ć ļ®öĒāĆņ£ĀņĀäņ▓┤ņØś ĻĖ░ļŖźņØ┤ ņ£äņĢö ļ░£ļ│æņŚÉ ņ¦üņĀæņĀüņ£╝ļĪ£ ļ»Ėņ╣śļŖö ņśüĒ¢źņØ┤ ĒÖĢņŗżĒ׳ ļ░ØĒśĆņ¦Ćņ¦ĆļŖö ņĢŖņĢśļŗż. ĒĢ£ Ļ░Ćņ¦Ć ņżæņÜöĒĢśĻ▓ī ĻĖ░ņ¢ĄĒĢĀ Ļ▓āņØĆ ņןņāüĒö╝ĒÖöņāØ ĒÖśņ×ÉņŚÉņä£ ņ”ØĻ░ĆĒĢśļŖö ļ®öĒāĆņ£ĀņĀäņ▓┤ ĻĖ░ļŖźņØ┤ļéś, Ēś╣ņØĆ ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ņĀ£ĻĘĀ ņ╣śļŻī Ēøä Ļ░ÉņåīĒĢśļŖö ļ®öĒāĆņ£ĀņĀäņ▓┤ ĻĖ░ļŖźņØ┤ ļ¬©ļæÉ ņ£äņĢöņØś ļ░£ļ│æņŚÉ ņśüĒ¢źņØä ļ»Ėņ╣śļŖö Ļ▓āņØĆ ņĢäļŗłļØ╝ļŖö ņĀÉņØ┤ļŗż. ļ░ĢĒģīļ”¼ņĢäņØś ņ£ĀņĀäņ▓┤ ļīĆļČĆļČäņØĆ ļ░ĢĒģīļ”¼ņĢä ņ×ÉņŗĀņØś ņāØņĪ┤Ļ│╝ ņ”ØņŗØņŚÉ ĒĢäņÜöĒĢ£ Ļ▓āņØ┤ņ¦Ć, ņłÖņŻ╝ņØś ņ£äņĢö ļ░£ļ│æņØä ņ£äĒĢ£ ļ¬®ņĀüņ£╝ļĪ£ ņĪ┤ņ×¼ĒĢśļŖö Ļ▓āņØĆ ņĢäļŗłĻĖ░ ļĢīļ¼ĖņØ┤ļŗż. ļö░ļØ╝ņä£, ņ£äņĢöņØś ļ░£ļ│æ ĻĖ░ņĀäņØä ĻĘ£ļ¬ģĒĢśĻ│Āņ×É ĒĢĀ ļĢī, ļŗ©ņł£Ē׳ ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ņĀ£ĻĘĀ ņ╣śļŻī ņĀä/Ēøä ļ│ĆĒÖöļ¤ēņØ┤ Ēü░ ļ®öĒāĆņ£ĀņĀäņ▓┤ ĻĖ░ļŖźņŚÉļ¦ī ņ┤łņĀÉņØä ļ¦×ņČöņ¢┤ ĒśäņāüņØä ņØ┤ĒĢ┤ĒĢśĻĖ░ļ│┤ļŗżļŖö, ņĀ£ĻĘĀ ņ╣śļŻī ņĀä/Ēøä ļ│ĆĒÖöļ¤ēņØ┤ Ēü¼ņ¦Ć ņĢŖļŹöļØ╝ļÅä ņ£äņĢö ļ░£ļ│æĻ│╝ ņŚ░Ļ┤Ćņä▒ņØ┤ ņ׳ņØä ņłś ņ׳ļŖö ņ£ĀņĀäņ×É ĻĖ░ļŖźņØä ņäĀļ│äĒĢśņŚ¼ ļČäņäØĒĢśļĀżļŖö ļģĖļĀźņØ┤ ĒĢäņÜöĒĢśļŗż.
ņ¦ĆĻĖłĻ╣īņ¦Ć ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ņĀ£ĻĘĀ ņ╣śļŻī ņĀä/Ēøä ļ»ĖņāØļ¼╝ĻĄ░ņ¦æņØś ļ│ĆĒÖö ņ¢æņāüņŚÉ ļīĆĒĢ┤ ņé┤ĒÄ┤ļ│┤ņĢśļŗż. ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ Ļ░ÉņŚ╝ ĒÖśņ×ÉņØś ņ£ä ļé┤ ļ»ĖņāØļ¼╝ĻĄ░ņ¦æņŚÉļŖö ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼Ļ░Ć ļ¦żņÜ░ ļåÆņØĆ ļ╣äņżæņØä ņ░©ņ¦ĆĒĢśĻ│Ā ņ׳Ļ│Ā ļ»ĖņāØļ¼╝ĻĄ░ņ¦æņØś ļŗżņ¢æņä▒ņØĆ ļé«ņĢäņĀĖ ņ׳ņ£╝ļ®░, ņĀ£ĻĘĀ ņ╣śļŻī Ēøä ļ»ĖņāØļ¼╝ĻĄ░ņ¦æņØś ļŗżņ¢æņä▒ņØĆ ņ”ØĻ░ĆĒĢ£ļŗż. ĻĘĖļ¤¼ļéś ņ£ä ļé┤ ļ»ĖņāØļ¼╝ĻĄ░ņ¦æņØ┤ ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ ņĀ£ĻĘĀ ņ╣śļŻīļź╝ ĒåĄĒĢ┤ ĒŚ¼ļ”¼ņĮöļ░ĢĒä░ ĒīīņØ╝ļĪ£ļ”¼ Ļ░ÉņŚ╝ ņĀäņ£╝ļĪ£ ņÖäņĀäĒ׳ ļ│ĄņøÉļÉĀ ņłś ņ׳ļŖöĻ░ĆņŚÉ ļīĆĒĢ┤ņä£ļŖö ļģ╝ļ×ĆņØ┤ ņ׳ņ£╝ļ®░, ņĀ£ĻĘĀ ņ╣śļŻīļź╝ ņŗ£Ē¢ēĒĢśņśĆļŹöļØ╝ļÅä ņ£äņČĢņä▒ ņ£äņŚ╝ņØ┤ļéś ņןņāüĒö╝ĒÖöņāØņØ┤ ļ░£ņāØĒĢ£ ĒÖśņ×ÉņØś ļ»ĖņāØļ¼╝ĻĄ░ņ¦æņØĆ ĻĘĖļĀćņ¦Ć ņĢŖņØĆ ĒÖśņ×ÉņØś ļ»ĖņāØļ¼╝ĻĄ░ņ¦æĻ│╝ ņ░©ņØ┤Ļ░Ć ņ׳ļŗż.
Fig.┬Ā1.
Microbial diversity and dysbiosis indices in gastric mucosal samples based on the data of the study by Park et al.[13] (A) Richness index, (B) Shannon index, and (C) microbial dysbiosis index. The healthy control indicates individuals with chronic superficial gastritis without Helicobacter pylori (H. pylori) infection. Disease control indicates individuals with severe atrophic gastritis and intestinal metaplasia without H. pylori infection. The microbial dysbiosis index has been calculated using the same formula reported by Guo et al.[15] It is depicted as follows: log (total abundance of genera decreased after successful eradication [i.e., Helicobacter] / total abundance of genera increased after successful eradication [17 genera demonstrated in the study by Guo et al.15]). To prevent the logarithm from becoming negative infinity, 0% of abundance in H. pylori is replaced by 0.1%. P-values are C calculated using the Mann-Whitney U test.
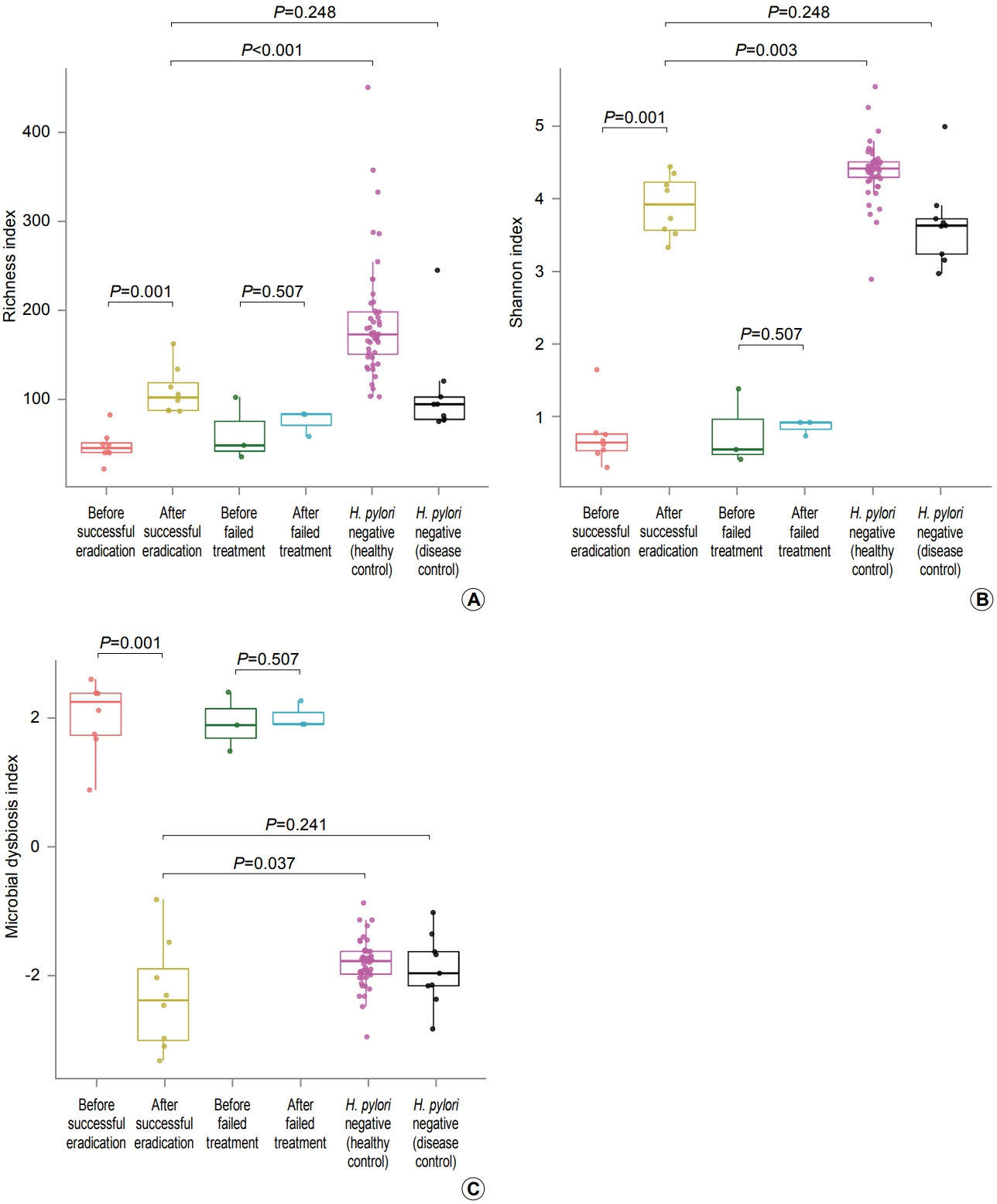
Fig.┬Ā2.
Principle component analysis for the gastric microbiome based on the data of the study by Park et al.[13] (A) Before vs. after successful Helicobacter pylori (H. pylori) eradication and (B) after successful H. pylori eradication vs. healthy control group vs. disease control group. Healthy control indicates individuals with chronic superficial gastritis without H. pylori infection. Disease control indicates individuals with severe atrophic gastritis and intestinal metaplasia without H. pylori infection. The chi-square transformed data matrix in the relative abundance at the family level has been subjected to the weighted linear regression analysis. ANOSIM, analysis of similarities.
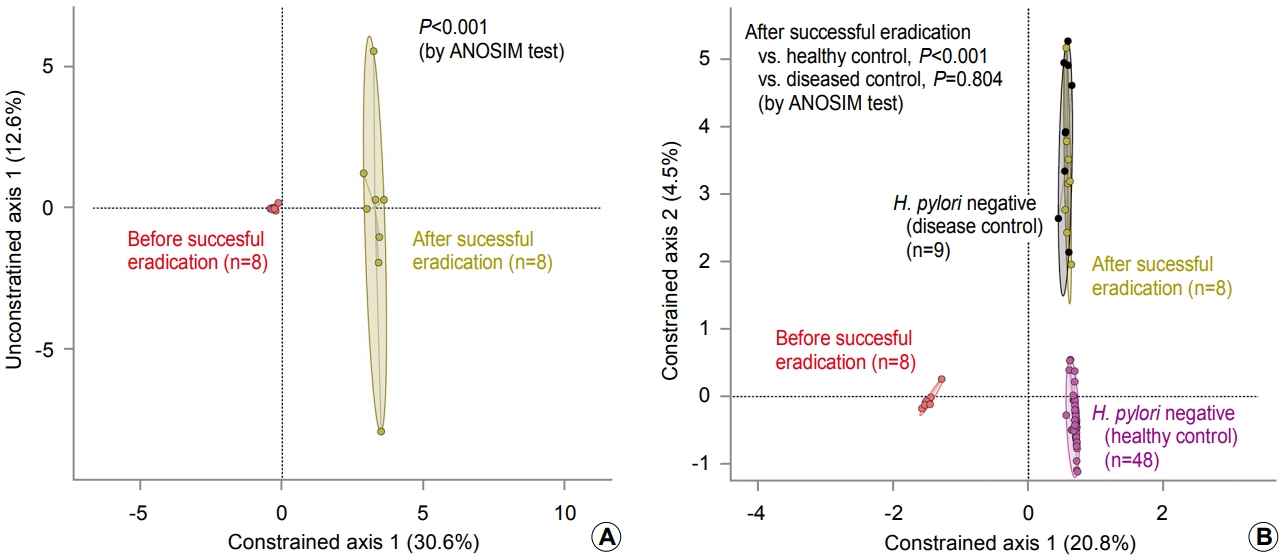
Fig.┬Ā3.
Schematic diagram of differences in the gastric microbiome according to the Helicobacter pylori (H. pylori) infection and eradication. aRestored bacterial taxa vary across the studies.
Table┬Ā1.
Summary of Studies on the Gastric Microbiome after Helicobacter pylori Eradication Therapy
| Study | Country | Number of patients | Population | Background disease and number of patients successfully treated with HP eradication therapy | HP eradication regimen | Location | Sample collection | Evaluation timing after HP eradication therapy | Microbiome analysis | Metagenomic function analysis | Main results |
|---|---|---|---|---|---|---|---|---|---|---|---|
| Li et al. [10] (2016) | China | 68 | Adult | Duodenal ulcer: 21 | Rabeprazole 40 mg, bismuth citrate, amoxicillin 2,000 mg, clarithromycin 1,000 mg, 10 days | Antrum and corpus | Biopsy | At least 4 weeks later | Quantitative real-time PCRforl6S rRNA gene | None | Antrum after HP eradication: Lactobacillus Ōåæ, Clostridium leptum Ōåæ, Enterobacteria Ōåæ, Clostridium coccoides Ōåæ |
| Corpus after HP eradication: Lactobacillus Ōåæ, Clostridium coccoides Ōåæ | |||||||||||
| Feces after HP eradication: Lactobacillus Ōåæ | |||||||||||
| Li et al. [11] (2017) | China | 33 | Adult | Gastritis: 7 | Esomeprazole 40 mg, amoxicillin 2,000 mg, clarithromycin 1,000 mg, 7 days | Antrum and corpus | Biopsy | 8 weeks later | 16S rRNA gene sequencing | None | Bacterial diversity after HP eradication Ōåæ |
| Intestinal metaplasia: 4 | OTUs after HP eradication: Neisseria Ōåæ, Serratia Ōåæ, Haemophilus parainfluenzae Ōåæ, Rhizobium Ōåæ, Flavobacterium Ōåæ, Fusobacterium Ōåæ | ||||||||||
| He et al. [12] (2019) | China | 17 | Adult | Chronic superficial gastritis: 10 | Esomeprazole 40 mg, bismuth subcitrate 440 mg, amoxicillin 2,000 mg, furazolidone 200 mg, 14 days | Antrum | Biopsy | 4 and 24 weeks later | 16S rRNA gene sequencing | PICRUSt | Bacterial diversity after HP eradication Ōåæ |
| At 24 weeks after HP eradication: Prevotella Ōåæ, Ralstonia Ōåæ, Pseudomonas Ōåæ, Lactobacillus Ōåæ | |||||||||||
| At 24 weeks vs. 4 weeks after HP eradication: Prevotella Ōåæ, Lactobacillus Ōåæ, Ralstonia Ōåō | |||||||||||
| Changes in metagenomic function after HP eradication: lipopolysaccharide biosynthesis Ōåō, lipopolysaccharide biosynthesis proteins Ōåō,bacterial motility proteins Ōåō,bacterial chemotaxis Ōåō, glycolysis/gluconeogenesis Ōåæ, glycine, serine, and threonine metabolism Ōåæ, pentose phosphate pathways Ōåæ | |||||||||||
| Park et al. [13] (2019) | Korea | 138 | Adult | Chronic superficial gastritis: 9 | Rabeprazole 40 mg, amoxicillin 2,000 mg, clarithromycin 1,000 mg, 14 days | Antrum | Biopsy | 1 month hter | 16S rRNA gene sequencing | PICRUSt | Before the HP eradication therapy, the relative abundance of Helicobacter was 93.3%, vdiile that of non-HP-Proteobacteria was 2.9%. After the eradication, the relative abundance of non-HP-Proteobacteria increased (46.8%). |
| Changes in microbial taxa and metagenomic function after HP eradication: Rhizobiales Ōåæ, T4SS genes Ōåæ | |||||||||||
| Serrano et al. [14] (2019) | Chile | 16 | Child | Children with nausea and abdominal discomfort: 7 | Omeprazole, amoxicillin, clarithromycin, 14 days | Stomach | Biopsy | 2 months later | 16S rRNA gene sequencing | None | Shannon and Simpson indexes after HP eradication Ōåæ, Pseudomo-nadaceae Ōåæ, Delftia Ōåæ, Kocuria Ōåæ, Stenotrophomonas Ōåæ |
| Guo et al. [15] (2020) | China | 164 | Adult | Normal or superficial gastritis: 5 | Omeprazole 40 mg, bismuth citrate 600 mg, metronidazole 1,200 mg, tetracycline 2,250 mg, 10 days | Antrum | Biopsy | 6 months later | 16S rRNA gene sequencing | Tax4Fun | Richness and Shannon indexes after HP eradication Ōåæ, Staphylococcus Ōåæ, Corynebacterium Ōåæ, Fusobaderium Ōåæ, Bacteroides Ōåæ, Streptophyta Ōåæ, Prevotella Ōåæ, Gemella Ōåæ, Porphyromonas Ōåæ, Alloprevotella Ōåæ, Veillonella Ōåæ, Neisseria Ōåæ, Streptococcus Ōåæ, Rothia Ōåæ, Haemophilus Ōåæ |
| Atrophic gastritis: 25 | Protein digestion and absorption Ōåæ, glycosaminoglycan and other glycan degradańżŠon Ōåæ, gastric acid secretion Ōåæ, carbohydrate digestion and absorption Ōåæ, flagellar assembly Ōåō, bacterial chemotaxis and bacterial secretion system pathways Ōåō | ||||||||||
| Intestinal metaplasia/dyspl asia: 28 | |||||||||||
| Miao et al. [16] (2020) | China | 55 | Child | Gastritis or duodenitis: 2 | PPI, amoxicillin, clarithromycin, 14 days, for 5 children | Antrum | Biopsy | Within 4 weeks later | 16S rRNA gene sequencing | PICRUSt | Microbial diversity after HP eradication Ōåæ |
| Gastric or duodenal erosion: 2 | PPI, bismuth, amoxicillin, clarithromycin, 14 days, for 3 children | Neisseria, Cuwibacter, and Haemophilus were common in HP-negative children and patients with successful HP eradication | |||||||||
| Duodenal ulcer: 7 | PPI, amoxicillin, 5 days, thereafter PPI, clarithromycin, 5 days (sequential therapy), for 2 children | ||||||||||
| Amoxicillin, clarithromycin, bismuth, 14 days, for 1 child | |||||||||||
| Sung et al. [6] (2020) | China | 202 | Adult | Gastritis, atrophy, or intestinal metaplasia: 102a | Omeprazole 40 mg, amoxicillin 2,000 mg, clarithromycin 1,000 mg, 7 days | Antrum and corpus | Biopsy | 1 year later | 16S rRNA gene sequencing | PICRUSt | Microbial diversity after HP eradication Ōåæ |
| Patients with persistent inflammation 1 year after HP eradication: Acinetobacter Iwoffii Ōåæ, Streptococcus anginosus Ōåæ, Ralstonia Ōåæ | |||||||||||
| Patients with emerged atrophy 1 year after HP eradication: Granulicatella Ōåæ, Streptococcus Ōåæ, Rothia Ōåæ, Leptotrichia Ōåæ | |||||||||||
| Patients with emerged intestinal metaplasia after HP eradication: Mesorhizobium Ōåæ, Cupriavidus Ōåæ | |||||||||||
| Changes in metagenomic function in patients with emerged intestinal metaplasia after HP eradication: amino acid metabolism Ōåæ, inositol phosphate metabolism Ōåæ, several genes encoding virulence bacterial T4SS Ōåæ |
REFERENCES
1. Marshall BJ, Warren JR. Unidentified curved bacilli in the stomach of patients with gastritis and peptic ulceration. Lancet 1984;1:1311ŌĆō1315.


3. Schistosomes, liver flukes and Helicobacter pylori. IARC Working Group on the evaluation of carcinogenic risks to humans. Lyon, 7-14 June 1994. IARC Monogr Eval Carcinog Risks Hum 1994;61:1ŌĆō241.


4. Karimi P, Islami F, Anandasabapathy S, Freedman ND, Kamangar F. Gastric cancer: descriptive epidemiology, risk factors, screening, and prevention. Cancer Epidemiol Biomarkers Prev 2014;23:700ŌĆō713.



5. de Assump├¦├Żo PP, Ara├║jo TMT, de Assump├¦├Żo PB, et al. Suicide journey of H. pylori through gastric carcinogenesis: the role of non-H. pylori microbiome and potential consequences for clinical practice. Eur J Clin Microbiol Infect Dis 2019;38:1591ŌĆō1597.


6. Sung JJY, Coker OO, Chu E, et al. Gastric microbes associated with gastric inflammation, atrophy and intestinal metaplasia 1 year after Helicobacter pylori eradication. Gut 2020;69:1572ŌĆō1580.
8. Eun CS, Kim BK, Han DS, et al. Differences in gastric mucosal microbiota profiling in patients with chronic gastritis, intestinal metaplasia, and gastric cancer using pyrosequencing methods. Helicobacter 2014;19:407ŌĆō416.


9. Jo HJ, Kim J, Kim N, et al. Analysis of gastric microbiota by pyrosequencing: minor role of bacteria other than Helicobacter pylori in the gastric carcinogenesis. Helicobacter 2016;21:364ŌĆō374.


10. Li L, Zhou X, Xiao S, Ye F, Zhang G. The effect of Helicobacter pylori eradication on the gastrointestinal microbiota in patients with duodenal ulcer. J Gastrointestin Liver Dis 2016;25:139ŌĆō146.


11. Li TH, Qin Y, Sham PC, Lau KS, Chu KM, Leung WK. Alterations in gastric microbiota after H. pylori eradication and in different histological stages of gastric carcinogenesis. Sci Rep 2017;7:44935.



12. He C, Peng C, Wang H, et al. The eradication of Helicobacter pylori restores rather than disturbs the gastrointestinal microbiota in asymptomatic young adults. Helicobacter 2019;24:e12590.


13. Park CH, Lee AR, Lee YR, Eun CS, Lee SK, Han DS. Evaluation of gastric microbiome and metagenomic function in patients with intestinal metaplasia using 16S rRNA gene sequencing. Helicobacter 2019;24:e12547.


14. Serrano CA, Pierre R, Van Der Pol WJ, Morrow CD, Smith PD, Harris PR. Eradication of Helicobacter pylori in children restores the structure of the gastric bacterial community to that of noninfected children. Gastroenterology 2019;157:1673ŌĆō1675.


15. Guo Y, Zhang Y, Gerhard M, et al. Effect of Helicobacter pylori on gastrointestinal microbiota: a population-based study in Linqu, a high-risk area of gastric cancer. Gut 2020;69:1598ŌĆō1607.
16. Miao R, Wan C, Wang Z. The relationship of gastric microbiota and Helicobacter pylori infection in pediatrics population. Helicobacter 2020;25:e12676.


17. Eckburg PB, Bik EM, Bernstein CN, et al. Diversity of the human intestinal microbial flora. Science 2005;308:1635ŌĆō1638.



18. Ott SJ, Musfeldt M, Wenderoth DF, et al. Reduction in diversity of the colonic mucosa associated bacterial microflora in patients with active inflammatory bowel disease. Gut 2004;53:685ŌĆō693.



19. Pflughoeft KJ, Versalovic J. Human microbiome in health and disease. Annu Rev Pathol 2012;7:99ŌĆō122.


20. Hansson LE, Nyr├®n O, Hsing AW, et al. The risk of stomach cancer in patients with gastric or duodenal ulcer disease. N Engl J Med 1996;335:242ŌĆō249.


21. Chua EG, Loke MF, Gunaletchumy SP, et al. The influence of modernization and disease on the gastric microbiome of orang asli, myanmars and modern malaysians. Microorganisms 2019;7:174.



-
METRICS

-
- 1 Crossref
- 3,433 View
- 116 Download
- Related articles in Korean J Helicobacter Up Gastrointest Res






